在当今生物医学研究中,实验手段和数据量正以前所未有的速度膨胀。从基因组学、单细胞组学到多模态数据,再到可穿戴设备的健康监测,科研人员每天都在与庞大的数据和复杂的分析流程打交道。
然而,实验设计琐碎、工具分散、跨学科整合困难,成为阻碍科研效率的重要瓶颈。
👉 想象一下,如果有一个 虚拟 AI 生物学家,能够自动理解研究问题,调用合适的数据库、工具和算法,甚至为你写好代码、执行分析,并输出结果报告------科研的效率会提升多少?
斯坦福 SNAP 团队推出的 Biomni,正是这样一位"科研助手"。它不仅能胜任跨领域的数据分析,还能为湿实验研究者设计实验流程,被称为首个 通用型生物医学 AI Agent。


一、Biomni 是什么?
Biomni 的目标,是构建一个 "虚拟 AI 生物学家",能像科研人员一样思考和执行任务,覆盖基因组学、免疫学、药理学、病理学、临床医学等多个领域。
架构组成
Biomni 包括两个核心部分:
- Biomni-E1:
一个统一的生物医学"动作空间",相当于实验室和计算环境的总和。它整合了:-
150 种专用生物医学工具
-
105 个常用生物信息学软件(支持 Python、R、Bash)
-
59 个数据库(如 PDB、ClinVar、OpenTarget、BindingDB 等) 并通过 AI 动作发现代理 从 2500 篇最新 bioRxiv 论文中自动提取研究步骤和工具。
-
-
Biomni-A1:
一个智能代理,能够调用 E1 环境。它通过 大语言模型 + 动态规划 + 代码执行,自动检索合适工具,制定执行计划,生成并运行代码,在执行过程中不断调整策略,保证灵活性与准确性。
换句话说,E1 提供实验室,A1 提供大脑。两者结合,使 Biomni 拥有跨学科的"科研执行力"。
二、Biomni 有何优势?
1. 覆盖面广,真正通用
Biomni 并不是为某个单一任务(如 scRNA-seq 分析)定制,而是能在 零样本(zero-shot) 情况下,处理多种复杂任务:
-
因果基因优先级排序
-
CRISPR 筛选设计
-
罕见病诊断
-
药物重利用
-
微生物组分析
-
分子克隆实验
2. 性能媲美甚至超过专家
在多个权威基准测试中,Biomni 表现突出:
-
LAB-Bench 数据库问答 (DbQA) :准确率 74.4%,与专家持平(74.7%),显著超过其他 AI(40.8%)。
-
序列问答 (SeqQA) :81.9%,高于专家(78.8%)。
-
HLE 跨 14 个学科测试:在陌生任务中,Biomni 得分 17.3%,远高于基础 LLM (6.0%) 和其他 AI agent (12.8%)。
-
平均性能提升 :比基础 LLM 高402.3%,比编码 Agent 高43%。
3. 自动化多步流程
在真实科研任务中,Biomni 平均每个任务执行 6--24 步,调用多个工具和数据库,自动组合成复杂的工作流,无需研究人员逐步硬编码。
4. 输出可视化报告
不同于只给结果的 LLM,Biomni 会生成:轨迹图、UMAP、热图、PCA 双标图、聚类图等,帮助科研人员快速洞察数据。
三、真实案例展示
案例 1:可穿戴传感器 + 多组学数据
研究者给 Biomni 提供了 485 份餐后体温数据(CGM 监测)、227 晚睡眠数据,以及 脂质组、代谢组、蛋白质组数据。
-
Biomni 自动分析后发现:
-
平均餐后体温上升 2.19°C;
-
睡眠效率在周三最高,周日最低,规律性比总时长更关键;
-
脂质、代谢物与蛋白质之间形成紧密调控网络。
这一过程,从原始数据到假设提出,完全自动完成。
案例 2:胚胎骨骼多组学数据
Biomni 处理了 33.6 万个胚胎细胞核的 snRNA-seq + snATAC-seq 数据,构建了 10 步分析流程:
-
比较不同解剖位置的细胞组成;
-
构建发育轨迹;
-
分析转录因子与靶基因调控网络。
它自动生成可视化结果,提出了与文献一致且可验证的新假设。
案例 3:分子克隆实验
研究者要求 Biomni 设计"将 B2M 基因的 sgRNA 插入 lentiCRISPR v2 载体"的实验。
-
Biomni 自动完成:
-
质粒结构解析
-
sgRNA 设计
-
寡核苷酸合成
-
Golden Gate 装配
-
菌落筛选与测序验证
-
实验人员严格按照 Biomni 的协议操作,成功获得正确克隆,验证其可靠性。
这意味着,Biomni 不只是"分析助手",还可以成为 湿实验研究的实验设计师。
四、Biomni 的意义
-
对科研人员:减少重复劳动,把更多精力放在假设生成和创新思考上;
-
对制药公司:可自动筛选靶点、设计干预实验、加速药物发现;
-
对临床:辅助罕见病诊断、基因优先排序,提升个性化医疗水平;
-
对公众健康:结合可穿戴设备,实现实时健康监测与干预。
五、未来展望与挑战
Biomni 虽然表现出色,但仍存在挑战:
-
对临床推理、创新实验设计等高层次任务仍不如专家;
-
倾向依赖最新文献,可能忽略经典方法;
-
尚未整合图像、病理切片等多模态信息。
未来,Biomni 团队计划:
-
引入强化学习机制,让代理能自我迭代提升;
-
更好地整合多模态数据(文本 + 图像 + 结构化信息);
-
自动发现并引入新工具与数据库,保持长远适用性。
📝 总结
Biomni = 实验室的 AI 科研助手
它能看文献、会写代码、懂组学,还能帮你设计实验。
无论你是生物信息学分析人员,还是实验室的分子生物学家,Biomni 都可能成为你未来工作中最强大的伙伴。
在人工智能推动科研的浪潮中,Biomni 代表着一个新起点------让 AI 真正成为科研过程中的 共同发现者。
-
官网网站 👉 https://biomni.stanford.edu
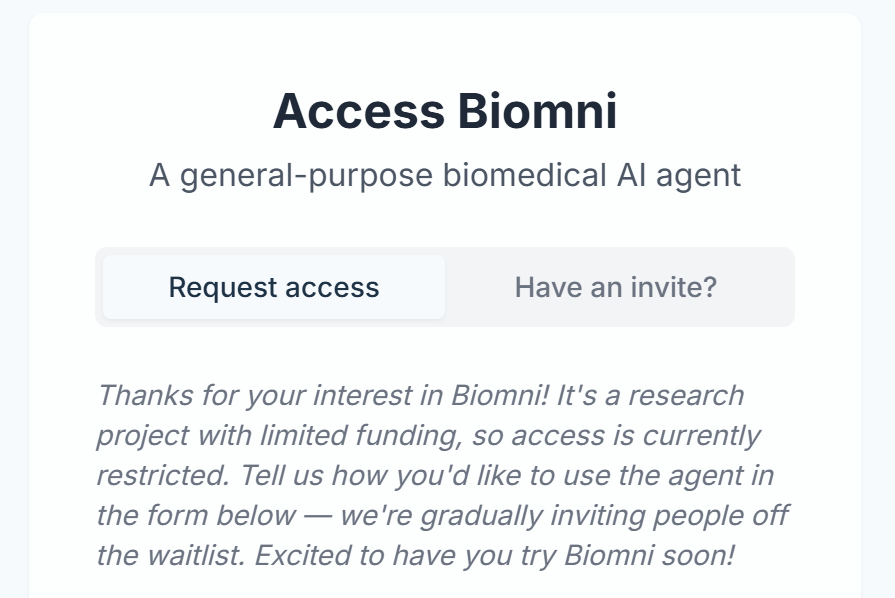
现在 Biomni 官网显示无法直接使用,是因为该项目还处于受控测试阶段。你可以通过 Request access 填写申请表,进入等待名单,等待官方邀请开放使用权限。
-
GitHub 开源地址 👉 https://github.com/snap-stanford/Biomni
Biomni 本地安装与配置
一、环境准备
-
操作系统:推荐使用 Ubuntu 22.04 (64-bit),已验证兼容性较好。
-
工具需求:安装 Conda(用于创建隔离环境),建议安装 Miniconda 或 Anaconda。
二、克隆仓库 & 环境搭建
-
克隆官方 GitHub 仓库:
git clone https://github.com/snap-stanford/Biomni.git cd Biomni -
执行设置脚本
setup.sh,构建基础环境:bash setup.sh此脚本将创建一个名为
biomni_e1的 Conda 环境,并安装所需依赖包。
> 注意:整个环境可能需要 约 10 小时 构建完成,并消耗 30 GB 存储空间。 -
环境搭建完成后,激活:
conda activate biomni_e1
三、安装 Python 包
-
从 PyPI 安装稳定版本:
pip install biomni --upgrade -
安装最新源码版本:
pip install git+https://github.com/snap-stanford/Biomni.git@main
四、配置 API Key 与环境变量
Biomni 依赖外部 LLM 模型(如 Claude、OpenAI、Gemini 等)。
方法一:通过 .env 文件配置
在项目根目录执行:
cp .env.example .env
编辑 .env 文件:
ANTHROPIC_API_KEY=你的_Claude_API_密钥# 可选字段
OPENAI_API_KEY=你的_OpenAI_密钥
OPENAI_ENDPOINT=https://... # Azure OpenAI 的 endpoint
GEMINI_API_KEY=你的_Gemini_密钥
GROQ_API_KEY=你的_groq_密钥
LLM_SOURCE=指定模型来源("OpenAI" / "Anthropic" / 等)BIOMNI_DATA_PATH=/path/to/your/data
BIOMNI_TIMEOUT_SECONDS=600
方法二:使用 Shell 环境变量
export ANTHROPIC_API_KEY="你的_API_密钥"
export OPENAI_API_KEY="你的_OpenAI_密钥"
export LLM_SOURCE="Anthropic"# AWS Bedrock 配置
export AWS_BEARER_TOKEN_BEDROCK="你的_密钥"
export AWS_REGION="us-east-1"
五、安全注意事项
Biomni 会以 系统权限执行 LLM 生成的代码,具备文件、网络、系统命令访问权限。
⚠️ 务必在隔离或沙箱环境中运行,避免潜在安全风险。
六:运行示例
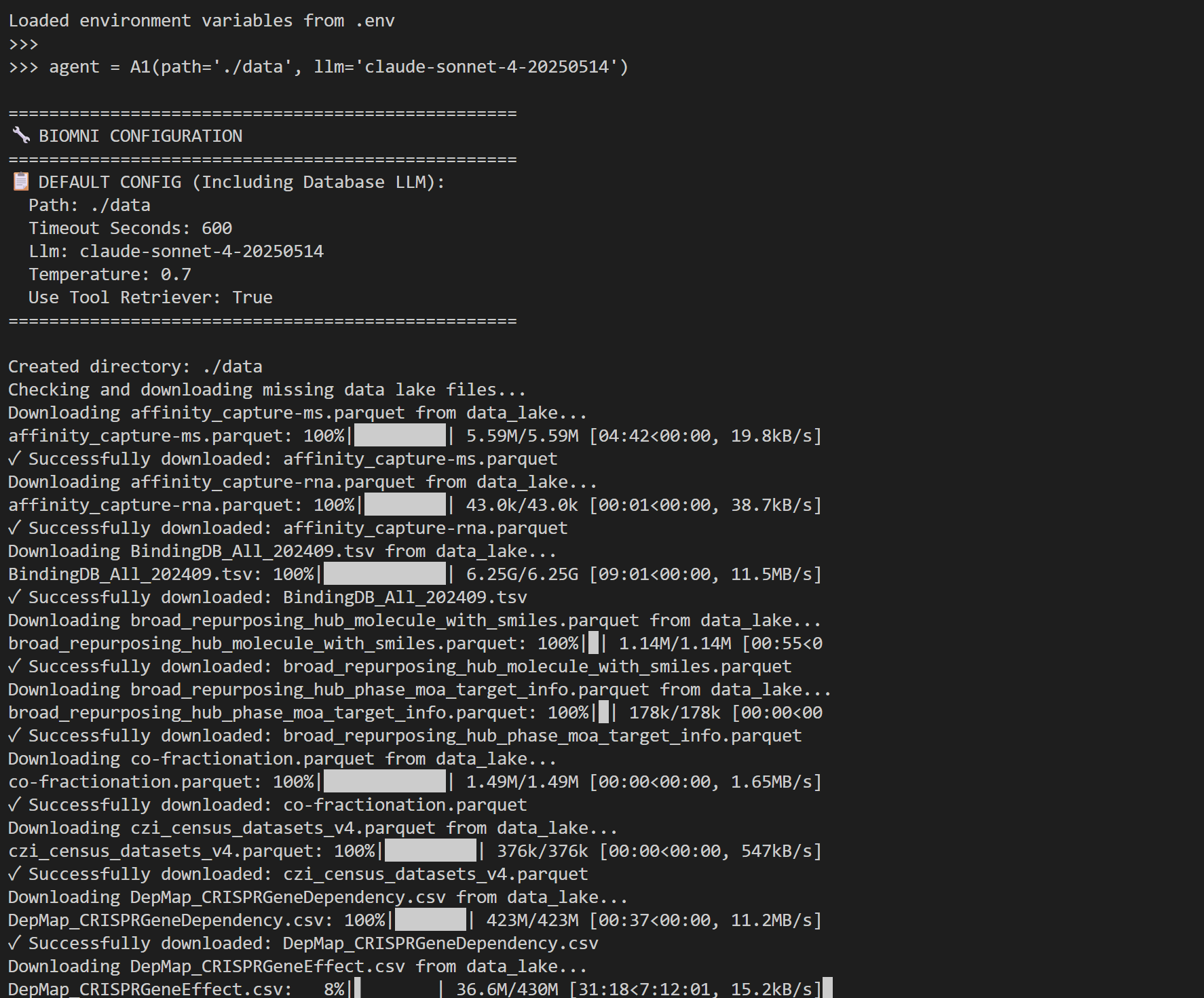
from biomni.agent import A1
agent = A1(path="./data", llm="claude-sonnet-4-20250514")
result = agent.go("Plan a CRISPR screen to identify genes that regulate T cell exhaustion, generate 32 genes…")
print(result)
使用前还要下载一个大概11G大小的文件,我的还在下载中,明天试试效果,给大家继续反馈。

(DAY 002))












![[网络入侵AI检测] 深度前馈神经网络(DNN)模型](http://pic.xiahunao.cn/[网络入侵AI检测] 深度前馈神经网络(DNN)模型)




)